Links是用来展现两个位置之间存在的联系;例如同源基因、两个国家等
主要的数据格式:
第一种格式是官方推荐的格式,两个位置放在同一行
而第二种格式在未来的版本可能会被淘汰:第一列表示links ID号,同一个ID号对应着两行

源代码如下:
- bezier_radius是描述link的弯曲程度;当为0时,绘画出直线
- record_limit = 2000最后再图形上只呈现2000条link
- data_out_of_range* = trim这是在links标签外设置的参数;从字面上可以理解为超出ideogram范围的数据过滤掉
关于links的geometry几何问题
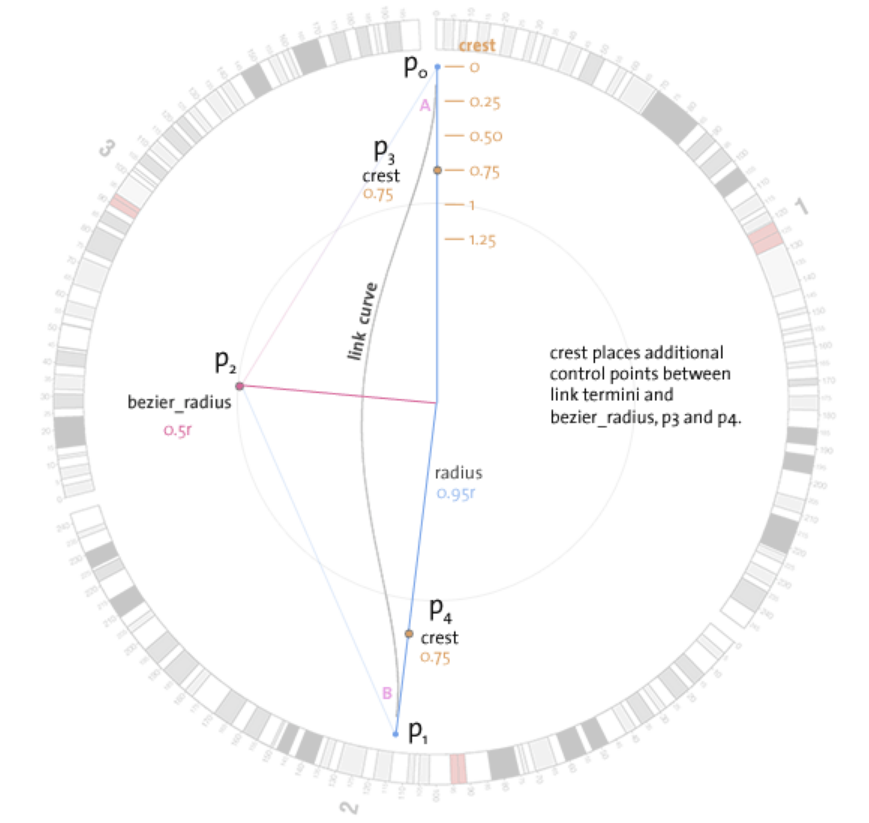
感觉这个要到具体美化的时候才会用到;先这样记着吧到时候再深入了解............
主要受到以下几个参数的控制
- bezier_radius控制曲率也就是p2点的位置
- radius控制连接处的位置也就是p0和p1位置
- crest则控制p3和p4点的位置;当crest为1时,p3和p4位于p2处;当crest=0时,p3和p4位于p1和p0处
当有多中links重叠时,使用z参数控制优先顺序是个不错的选择

放大图片也可以看到绿色是画在红色上面的!这也就是z参数的作用
在每个单独的link标签内部还可以分别设置对应的bezier_radisu和crest以及link等自定义参数
关于links有一些很实用的rule
on(CHR)returns 1 if data point on chromosomeCHR, for links tests both endsfrom(CHR)returns 1 if start of link is on chromosomeCHR, used for links onlyto(CHR)returns 1 if start of link is on chromosomeCHR, used for links onlybetween(CHR1,CHR2)returns 1 if link is betweenCHR1andCHR2, regardless of orientation, used for links onlyfromto(CHR1,CHR2)returns 1 if link is fromCHR1toCHR2, used for links onlytofrom(CHR1,CHR2)returns 1 if link is fromCHR2toCHR1, used for links only
on(CHR,START,END)returns 1 if data point is on chromosome CHR and intersects coordinate START-END, for links tests both ends
within(CHR,START,END)returns 1 if data point is on chromosome CHR and falls entirely within START-END, for links tests both endsabs(var(pos1) - var(pos2))< 100Mb这个主要是应用在condition = var(intrachr)染色体内部的链接规则;当两个连接点的绝对距离满足规则就执行-----

这个图我用了3个规则
关于links一些属性的设置同样可以应用rule规则
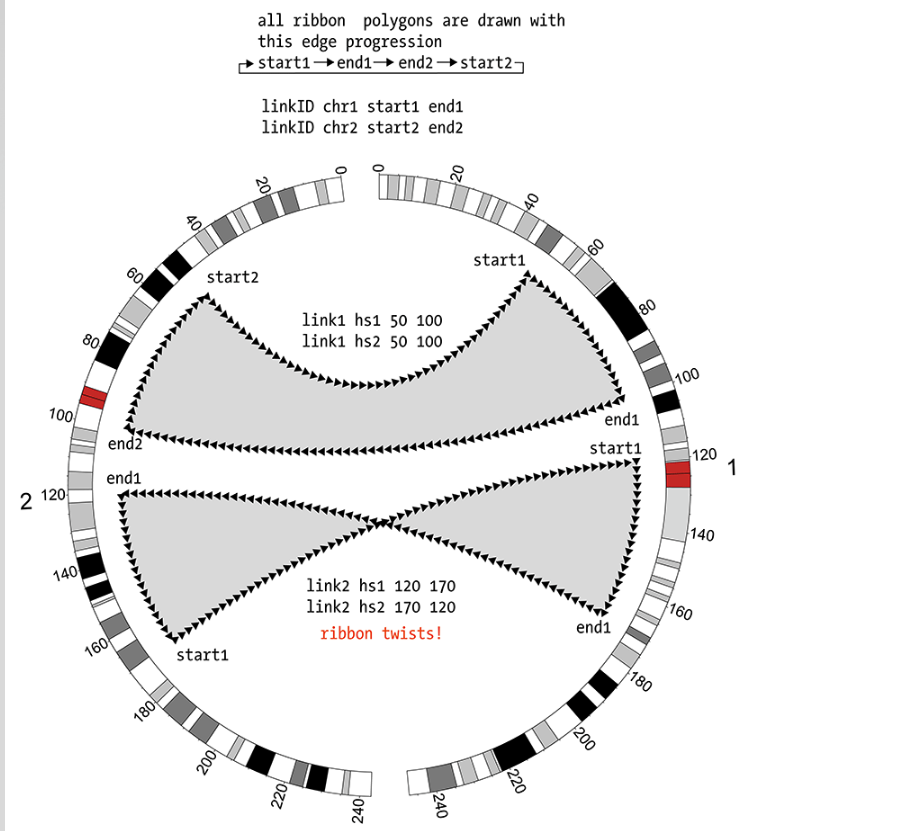
最后就是关于ribbon的展示了;当links的区域足够大时就可以使用ribbon进行展示
ribbon=yes将显示绸带状的links
stroke_color&&stroke_thickness设置边框
ribbon怎么让它变得扭曲;加上twist参数,强制扭曲
links内容就学习到这里啦!有些部分感觉很简单就没有直接实践,从官网直接截的图方便理解!